Le mésocentre de calcul MesoBFC est une plateforme scientifique d’appui à la recherche pour les chercheurs spécialistes en simulations numériques haute performance et en modélisations dans différents domaines scientifiques, (physique, chimie, biologie, climatologie …) ou dans le domaine médical.
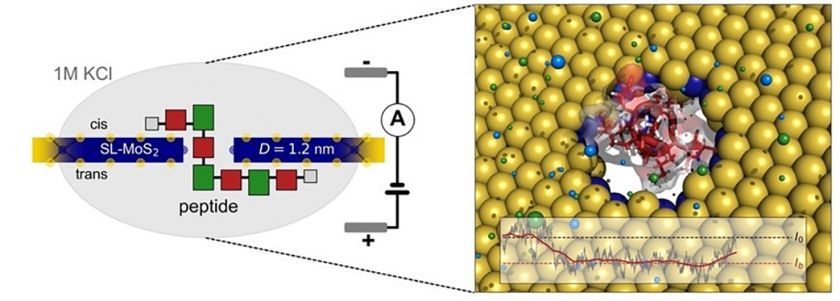
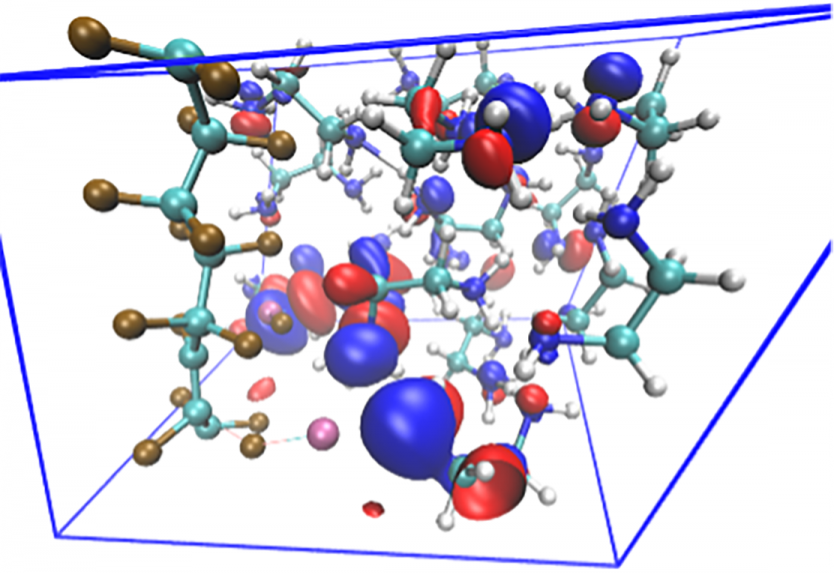
Cet appel à projet MesoBFC-Defi CPU a permis à 5 chercheurs de l’Université Marie et Louis Pasteur, de l’Université Bourgogne Europe et de l’Institut Agro Dijon, de bénéficier d’un supercalculateur composé de 48 serveurs de calcul pour un total de 2304 cœurs pour développer 4 projets scientifiques dans les domaines de la chimie, la biophysique et la climatologie. Ce sont plus de 4 millions d’heures de calcul, pour le calcul distribué de type MPI *, pendant une durée d’un mois qui ont permis aujourd’hui à ces projets d’apporter des avancées scientifiques majeures dans leur domaine.
Des avancées dans le domaine de la biophysique :
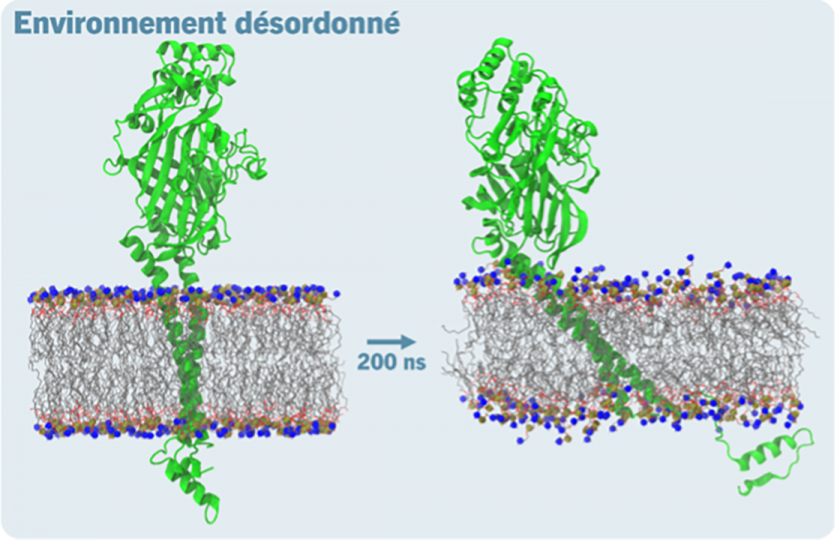
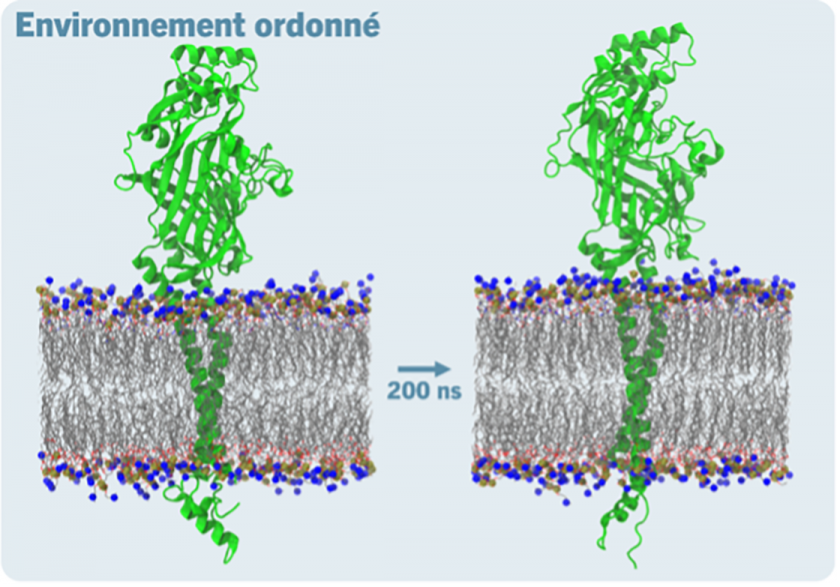
Le projet DECLIP sur lequel travaille le chercheur Florentin ALLEMAND du laboratoire Chrono-Environnement de l’Université Marie et Louis Pasteur, pourrait aider à mieux traiter les malades du cancer de manière plus ciblée.
En effet, l’objectif de ce projet est d’utiliser les lipoprotéines de haute densité (HDL), présentes naturellement dans le sang, pour transmettre des molécules à visée thérapeutique aux cellules malades. Les lipoprotéines dans le sang servent habituellement à transporter le cholestérol des tissus périphériques vers le foie afin de l’éliminer. Le défi MesoBFC a permis de démarrer ce projet ambitieux grâce aux grands moyens numériques mis à disposition. Des simulations de dynamique moléculaire ont été menées pendant cette campagne de calcul intensif.